Genome Biology|结构变异分析揭示绵羊与山羊进化中的趋同特征
标题:
Structural variant landscapes reveal convergent signatures of evolution in sheep and goats
期刊:
Genome Biology(12.3)
发表单位:
中国农业大学
研究背景:
家畜(如绵羊和山羊)在驯化过程中与野生祖先发生了显著的表型转变,称为“驯化综合征”。尽管这种转变明显,但驱动家畜趋同进化的遗传机制尚不完全清楚。结构变异(SVs)作为基因组的重要组成部分,对牲畜的进化、适应性和表型变异有重要影响。然而,SVs及其功能的全基因组表征在家畜中仍较缺乏。鉴于绵羊(Ovis aries)和山羊(Capra hircus)具有相似的驯化历史和选择压力,它们成为研究近亲家养物种趋同进化的理想模型。
研究材料:
1.绵羊材料:7种野生绵羊的37个样本和129个驯化种群(包括95个地方品种和34个改良品种)的495个样本。
2.山羊材料:6种野生山羊的72个样本和37个驯化种群(28个地方品种、6个改良品种和3个未分类种群)的209个样本。
3.古山羊材料:来自亚洲、欧洲和中东地区的古山羊遗骸的基因组数据。
研究方法:
测序方案:
群体测序方案:二代Illmina WGS测序:~15×(古山羊0.001-3.90×)
基因组组装测序方案:PacBio Sequel + Illumina NovaSeq + HiC + BioNano测序
ATAC-seq:66份绵羊和5份山羊材料不同组织部位的样本进行ATAC-seq
RNA-seq:24份绵羊和8份山羊材料不同组织部位的样本进行RNA-seq
分析方案:
1.基因组从头组装及完整性评估
2.基因组重复序列和非编码RNA预测
3.基因组注释
4.富集分析:KEGG富集、GO富集
5.SVs检测与热点分析
6.遗传多样性分析:核苷酸多样性分析、连锁不平衡分析、杂合度分析
7.群体结构分析:PCA分析、聚类分析、admixture分析
8.选择进化分析:Fst分析、DI分析、PBS分析
9.全基因组关联分析
10.开放区域peak分析
主要研究结果:
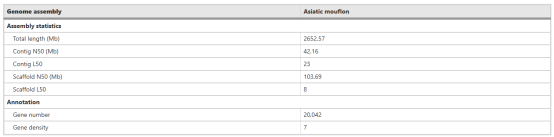
亚洲mouflon基因组的从头组装和注释
作者利用PacBio、Illumina、BioNano和HiC数据进行多轮组装,最终获得了亚洲mouflon染色体水平基因组Amuf_v1,总大小为2.65 Gb,contig N50长度为42.16 Mb,scaffold N50长度为103.69 Mb,包含27条44.04-282.18 Mb的染色体(表1)。在基因组完整性上,BUSCO分析显示,98.57%的真核生物通用基因在Amuf_v1中得到了覆盖。reads回比率和NGS数据覆盖率分别为99.89%和99.73%。此外,kmer分析和BLAST比对结果也揭示了93.15%的kmer完整性和99.71%的后生动物序列比对率。
在Amuf_v1基因组中,作者根据基因结构预测了20,042个基因,其中18,790个(93.75%)被功能注释,平均基因长度是47.09 kb。Amuf_v1基因组的高质量组装和详细注释为理解该物种的遗传特性与进化关系提供了坚实基础。
SVs的发现和表征
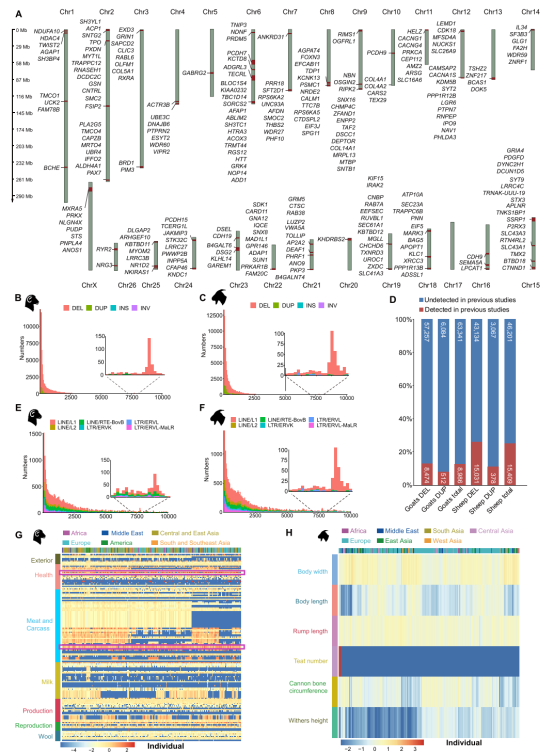
532份绵羊样本、281份山羊样本和84个古山羊样本平均测序深度分别为18.32×、21.45×和0.84×。利用2种及以上的软件进行SVs检测,绵羊和山羊分别鉴定到72,883个和86,283个长度在50bp - 1Mb的SVs(图2)。在不同的分类中,野生种群比本地种群和改良种群有更多的SVs,而且本地种群和改良种群间有更多共有SVs(图2A)。不同类型的SVs中,缺失是绵羊和山羊中最常见的类型(图2C-F)。SVs频率分布偏于稀有等位基因,绵羊和山羊中分别有26,157个(35.89%)和28,160个(32.51%)SVs的次要等位基因频率(MAF)< 0.01(图2D)。在SVs长度分布上,大多数SVs小于1 kb(图2C)。

图2 SVs集合的特征
SVs热点的分布
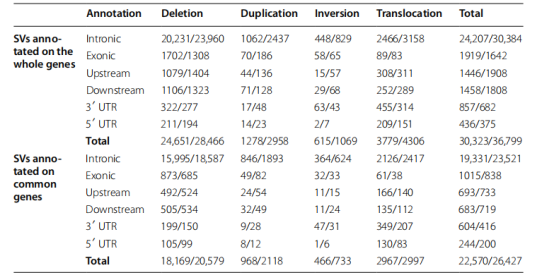
根据SVs断点在基因组位置,在绵羊和山羊染色体上发现了260个191个SVs热点,分别影响了1547个基因和1591个基因(图3A)。通过将热点与已知QTL进行比较,作者发现绵羊产奶量和腰肉产量等产量性状的120个热点与401个QTL重叠,山羊体长、乳房深度、乳头数量、骨质量和乳头位置等7个热点与7个QTL重叠。进一步分析发现,绵羊和山羊端粒中的SVs断点均显著富集。
SVs相关基因和转座因子相关SVs
作者对SVs进行注释,结果表明大多数SVs分布在基因间区,其次是内含子区,最后是外显子或者上游区域(表2)。通过比对SVs基因(与SVs重叠的基因)与QTL区域,作者发现绵羊和山羊的SVs分别与342个和7个QTL重叠,这些QTL与体重、胴体、纤维、体型等性状相关。此外,对不同QTL中SVs的富集分析发现,绵羊的SVs主要富集于1个疾病相关QTL和2个肌肉相关QTL,山羊的SVs主要富集于2个形态性状QTL(图3G、H),表明SV基因在生产性状中具有潜在的重要作用。转座元件分析显示,与SVs相关的最丰富TE家族包括LINEs的L1、L2和RTE-BovB,SINEs的tRNA-Core-RTE、Core-RTE和MIR,LTRs的ERV1-MALR、ERV1和ERVK,以及DNA转座子的hAT-Charlie(图3E、F)。在山羊和绵羊中,长度为100 - 150 bp和7500 - 8000 bp的SVs和TE数量均增加,可能是tRNA-Core-RTE和L1/RTE-BovB家族造成SVs数量增加(图3B、C、E、F)。此外,将已发表的SVs与检测到的SVs进行比较,作者发现绵羊中74.99%的复制缺失和山羊中87.58%的复制缺失是新生SVs(图3D)。
遗传多样性与群体结构
LD分析结果显示绵羊和山羊之间的LD衰退模式相似,野生种的LD衰退率最低且LD水平最高,其次是本地和改良种群(图4A、B)。驯化绵羊(1.32e−06)和山羊(1.32e−06)的核苷酸多样性(π)接近其野生祖先亚洲mouflon(1.55e−06)和bezoar山羊(1.00e−06)(图4C、D)。驯化绵羊的杂合度值(0.098)低于野生祖先亚洲mouflon(0.111),但驯化山羊的杂合度值(0.078)高于 bezoar山羊(0.045)(图4E、F)。基于SVs计算的Fst值在野生绵羊与驯化绵羊之间为0.06 - 0.87,野生山羊与驯化山羊之间为0.06 - 0.77(图4G、H)。较低的Fst值(驯化绵羊与亚洲祖先野山羊为0.12;驯化山羊与祖先野山羊为0.06),表明它们具有较近的系统发育关系。

图4 基于SVs绵羊和山羊样本的遗传多样性
在控制缺失基因型后,绵羊和山羊分别保留47,092个和58,279个SVs用于群体遗传结构分析。对532只现代绵羊和281只现代山羊的PCA分析显示,驯化绵羊与亚洲野生祖先moufon和欧洲野生祖先moufon的关系更为接近,驯化山羊与野山羊祖先bezoar及markhor的关系也较为紧密(图5B、C)。admixture分析表明野生种和亚洲、非洲、欧洲的驯化绵羊(K=5)和山羊(K=6)形成了不同的簇(图5D、E),与PCA结果一致。此外,系统发育树显示驯化绵羊和山羊种群分为来自不同大陆的三大群体,而野生种位于驯化种群之外,非洲绵羊被分为两个谱系(图5F、G)。
图5 绵羊和山羊的群体结构
针对重要农艺性状选择的候选SV基因
为了识别驯化过程中与重要农艺性状相关的SVs及其基因,作者比较了不同表型的驯化群体之间的PBS估计值。针对繁殖能力,繁殖和非繁殖绵羊群体之间PBS值前5%的SVs与403个基因重叠,这些基因在神经系统和催产素作用中发挥重要作用,如ADCY8和BMPR1B(图6B)。在山羊中,282个与繁殖表型相关的SVs基因显示出与动物器官发育和神经系统相关的功能(图6C),如BMPR2。共检测到19个在绵羊和山羊中有趋同选择的基因,这些基因对卵泡生长和胎次大小至关重要。此外,作者识别了绵羊和山羊中羊毛细度、乳制品和肉类性状的272、241、287个和205、230、261个SVs基因,其中部分基因显示出趋同选择信号(图6D),这些基因在相关性状的调节中发挥了重要作用。

图6 SVs尺度下绵羊和山羊的趋同进化概述
在趋同选择下功能基因的分子平行性
通过整合共同候选基因,作者发现79个同源基因在绵羊和山羊中经历了趋同选择,显著高于随机预期(P < 0.001)(图6E),这些基因占绵羊和山羊所有候选选择基因的5.07%和7.29%,并且显著富集于与繁殖相关的通路,如长期抑制、雌激素信号、催产素信号、Hippo信号和TGF-beta信号通路(图6F、G)。这些通路调节雌激素生产和繁殖特征,影响卵母细胞极性、减数分裂和胚胎发育(图6F、G)。在繁殖性状选择的基因中,有三种趋同选择的同源基因(BMPR1B、ADCY3和GRID2)涉及这些通路,而其他基因如PLCB1也显示出在生殖系统中的关键功能(图6G)。
BMPR1B 和 BMPR2 缺失的分子分析
通过对繁殖性状进行GWAS分析得到一系列候选基因(图7C、D),其中BMPR1B和BMPR2还是趋同选择的基因(图6B、C)。进一步研究发现BMPR1B的两种缺失变异(DEL00034481和DEL00067921)分别影响绵羊和山羊的繁殖力。BMPR1B附近核苷酸多样性分析显示,与低产群体相较,高产绵羊种群中DEL00034481的区域核苷酸多样性降低,而山羊群体中DEL00067921区域的核苷酸多样性则有所增加(图7A、B)。而且这两个SVs的等位基因频率在高产和非高产种群中存在显著差异(图7E、F)。
对DEL00034481序列进行转录因子结合基序(motif)预测,结果表明预测的2个motif与嗅觉、胚胎发育相关(图7H)。而且DEL00034481与报道的因果SNP不存在连锁,因此作者推测DEL00034481可能引入了motify,增加了绵羊卵巢等生殖组织中BMPR1B的表达(图8B),与不受SNP影响的多产绵羊的高生育能力有关。另一序列DEL00067921在山羊BMPR1B中存在,其预测的motif与生长、发育相关。在山羊中,BMPR1B在卵巢卵泡中表达较高(图8B),而DEL00067921的缺失未与选定的SNP连锁(图7G),同样说明DEL00067921影响转录因子结合降低BMPR1B表达,进而与山羊的多产性相关。
BMPR2在驯化中也经历了趋同选择,绵羊和山羊中分别发现2个缺失变异显示选择特征。这些SVs与BMPR1B中观察到的模式相似,且BMPR2中SVs可能独立于SNP影响生育力。研究还发现BMPR2缺失可能位于上游的增强子区域,形成BMPR2在生殖组织中的高表达(图8A)。

图7 BMPR1B基因缺失的进化与功能分析

图8 SVs对调控元件的影响
SVs对调控元件的影响
66份绵羊和5份山羊的ATAC-seq和SVs综合分析显示,绵羊和山羊的外显子及调控区域Peak-SVs比例较高,表明SVs对这些区域开放染色质影响显著(图8C、D)。利用ATAC-seq数据,作者在绵羊基因组中鉴定出646,121个潜在增强子,其中75,449个与SVs重叠,被认为是绵羊基因组中最可能的增强子区域,为理解SVs对调控元件的影响提供了新视角。





