Horticulture Research|无端粒间隙的牛油果全基因组参考序列组装为识别脂肪酸生物合成及抗病相关基因提供宝贵资源
标题:
期刊:
发表单位:
中国科学院大学昆明生命科学学院、云南大学生命科学学院
研究背景:
牛油果(Persea americana Mill)作为热带常绿植物,富含营养,其经济价值显著,但生产受病虫害困扰。研究聚焦于其抗病基因NLR(leucine-rich repeat receptor)及脂肪酸合成途径,探索提升品质之路。借助第三代测序技术,实现了牛油果的无间隙T2T基因组组装,精确覆盖端粒、着丝粒等复杂区域,填补了研究空白。本研究进一步分析了牛油果各组织中抗病与脂肪酸合成基因的表达,为理解其生物学特性及遗传改良奠定了坚实基础。T2T基因组的完成,将加速牛油果品种改良与产业升级。
来自一棵牛油果树的叶、茎和果实样本。
研究方法:
测序方案:
二代BGI WGS测序:51.9 Gb(60.1×)
二代Illumina RNA测序:叶、茎和果实不同组织部位的样本
三代PacBio HIFI测序:70.9 Gb(82.1×)
三代ONT测序:39.3 Gb(45.5 ×)
Pore-C测序:89.8 Gb(104.0×)
分析方案:
1.基因组组装及注释
2.基因组组装质量评估:比对率、覆盖深度、基因密度和重复序列密度、QV值、GC含量、LAI、BUSCO
3.着丝粒区域与结构变异区域识别
4.NLR基因识别和转录组分析
5.荧光原位杂交
主要研究结果:
通过NGS数据进行survey,确定了基因组大小(864 Mb)和杂合率(0.637%)。随后,利用PacBio HiFi、ONT ultra long和Pore-C测序数据对基因组进行组装和挂载得到初步组装的基因,然后利用ONT和HIFI数据对初步组装基因组进行补洞和端粒区域填充,最终的牛油果基因组大小为841.6 Mb,包括12条无间隙染色体和24个端粒,N50为78.8 Mb(图1A)。
对基因组进行质量评估,HiFi reads、ONT reads、NGS reads和 RNA-seq reads的整体比对率为 99.55%、99.91%、97.86%和99.1%。QV值、LAI值和BUSCO分别为56.23、15.99和99.4%(图1A)。
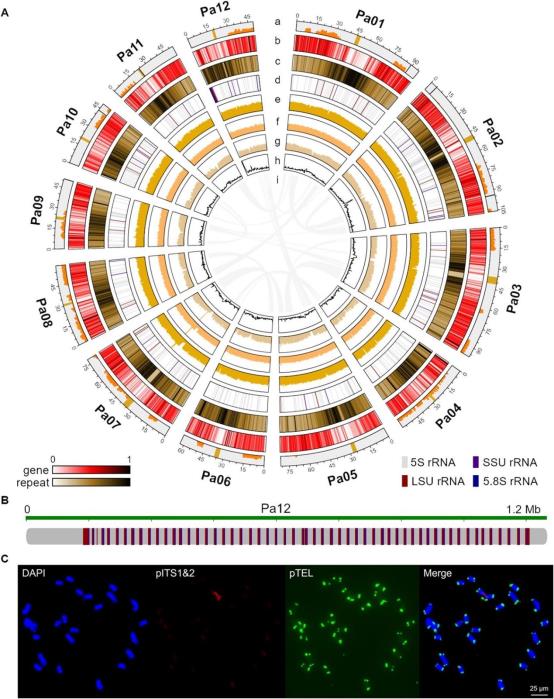
图1端粒到端粒无间隙牛油果基因组组装的景观
基因组注释
基于RNA-seq数据、同源蛋白和从头预测的结果作者获得了40,629个蛋白质编码基因,它们以对称的模式分布在两条染色体臂上,而重复序列则集中在相对中心的区域(图1A)。将上述基因在NR、Swiss-Prot、Pfam、GO和KEGG数据库进行功能注释,分别得到32,645、23,485、24,877、13,977和13,786个蛋白质。此外,通过HiFi reads重新比对,鉴定出大量杂合位点,主要位于基因间区和内含子区(图1A)。
非编码RNA也被预测并分类。特别地,Pa12染色体上的NOR区域包含多个45S rDNA单元,由一组小亚基rRNA、ITS1(internal transcribed spacer1)、5.8S rRNA、ITS2和首尾相连的大亚基rRNA组成(图1B),且Pa12末端GC含量高(图1A)。此外作者还根据组装的基因组设计了FISH探针,验证了NOR和端粒的真实性(图1C)。
牛油果着丝粒特征
利用迭代识别和聚类方式识别12条染色体上着丝粒的位置,并识别12个CSCR(chromosome-specific centromeric repeats)区域(图2A)。其中七种CSCR具有相似的序列,总是以头尾相接的方式出现在相应的着丝粒上(图2A),这些CSCR构成了SCG(Seven CSCRs Group)(图2B)。而在非SCG染色体上,CSCR04、CSCR11和CSCR12则以间隔的方式排列,而CSCR09和CSCR10在相应染色体上则相对罕见(图2A)。为了验证CSCR的真实性,我们基于共同的CSCR序列设计了FISH探针,证实了这些CSCR的存在(图2B)。
研究还发现,着丝粒两侧各1 Mb的区域包含了CSCR以及卫星DNA和TEs。LTR/Gypsy富集区与SCG富集区之间存在显著的重叠,而非SCG着丝粒则包含多种类型的TEs(图2A)。将这些CSCR与重复序列比对后,结果发现这些CSCR与多种TEs之间存在显著的相似性,推测TE的插入可能塑造了牛油果的着丝粒结构。
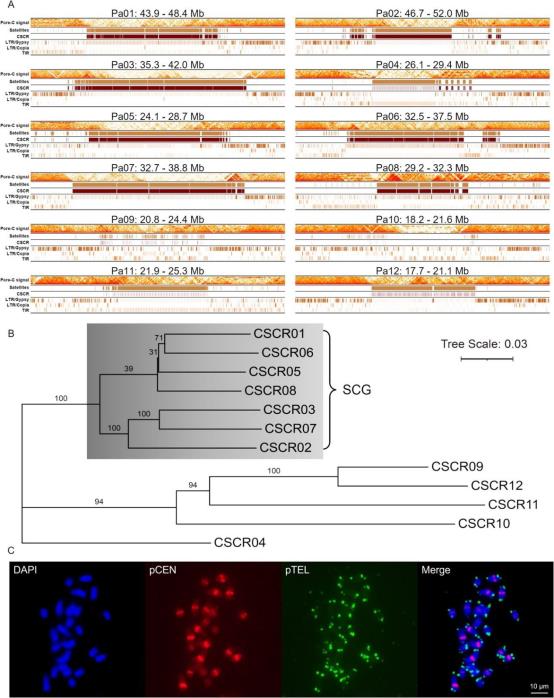
图2牛油果的着丝粒结构
结构变异分析
为了筛选Hass牛油果和西印度牛油果基因组之间的差异,作者分析了它们的结构变异。大规模的结构重排主要发生在复杂的着丝点区域附近,如Pa02上的易位和Pa12上的倒位(图3)。共鉴定出582,485条插入/缺失(InDels),其中7,668条插入和7,685条缺失长度超过50 bp。
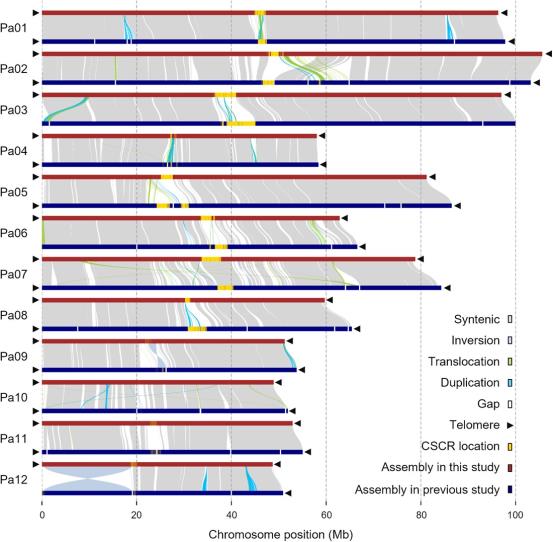
图3 牛油果组装之间的结构变异
探索牛油果NLR基因
作者在牛油果的西印度和Hass基因组中鉴定了分别为376个和230个主要NLR基因,这些基因分为三类亚家族:CNL(Coiled-Coil NB-ARC Leucine-rich-repeat)、TNL(Toll/interleukin-1 receptor NB-ARC Leucine-rich-repeat)和RNL(Resistance to Powdery Mildew Locus 8 NB-ARC Leucine-rich-repeat),其中CNL亚家族占96.54%(图4B)。NLR基因成簇状分布在整个基因组中(图4A),利用NLR基因编码的蛋白序列构建了进化树(图4B),许多染色体上物理位置接近的NLR基因聚集在一起,反映了它们密切的系统发育关系(图4A, B)。DupGen finder结果表明,这些基因可能起源于基因复制事件。
作者还分析了不同组织部位的NLR基因表达,结果发现NLR基因在茎中的总体相对表达水平高于叶片和果实(图4C),但部分基因在三种组织中均高表达(如Pa11g0262),推测这些基因可能参与所有植物组织的疾病反应。
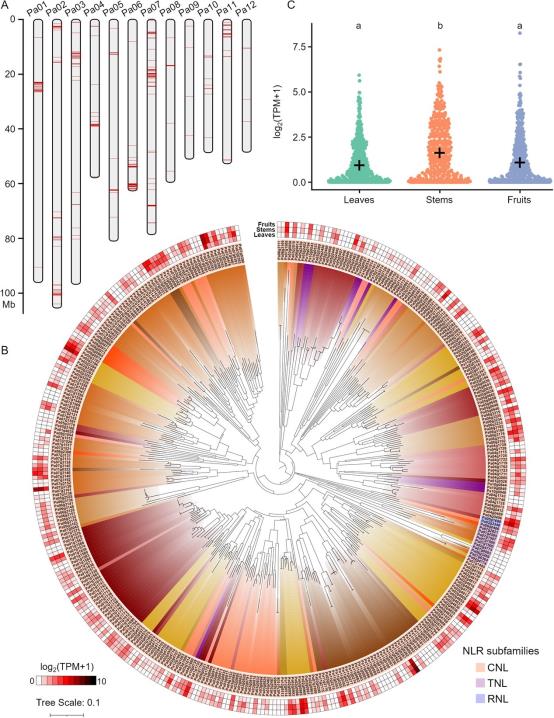
图4牛油果NLR基因的系统发育和转录组分析
脂肪酸生物合成途径基因的表达分析
脂肪酸含量是牛油果品质的关键。通过基因分析,作者发现128个与脂肪酸合成相关的基因,其中48个涉及质体中的从头合成,80个参与内质网中的三酰甘油(TAG)形成(图5)。关键酶丙酮酸脱氢酶(PDH)、乙酰辅酶A羧化酶(ACCase)和丙二酰辅酶A:酰基载体蛋白转酰基酶(MCMT)对质体内丙二酰CoA合成至关重要,且编码它们的基因在果实中高表达(图5)。编码酮酰-ACP合酶(KAS III)的脂肪酸合成基因如Pa08g1910在果实中的表达远超叶片和茎,表明其在果实脂肪酸积累中的重要作用。而编码硬脂酰-ACP脱饱和酶(SADs)的基因Pa02g0113在叶片中高表达,促进C18不饱和脂肪酸的合成。在TAG形成阶段,FAD2基因Pa07g1095、Pa07g1091和Pa12g0002特异性表达于果实,影响不饱和脂肪酸含量。综上,果实中高表达或特异性表达的基因显著影响牛油果的脂肪酸组成和含量。
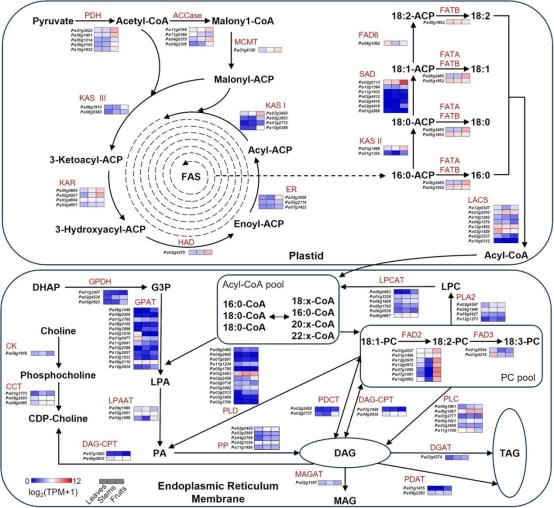
图5脂肪酸生物合成途径相关基因分析



