NC|拥有广泛基因组重排的蝴蝶中三维染色质结构的进化模式及其功能效应
Evolutionary patterns and functional effects of 3D chromatin structures in butterflies with extensive genome rearrangements
期刊:
Nature Communications(16.6)
发表单位:
西北工业大学、中国科学院
研究背景:
自达尔文时代起,蝴蝶的多样形态特征一直是生态学和进化生物学的重要研究对象。先前研究显示,凤蝶科蝴蝶多具有2n=60的核型,但青凤蝶属(Graphium)蝴蝶却存在显著核型变异,表明其经历了广泛的染色体重排,且基因组远大于其他蝴蝶。同时,脊椎动物中染色体间的三维染色质相互作用罕见,而哺乳动物中染色体重排可致三维染色质改变,影响进化与适应。然而,昆虫中三维染色质结构的跨物种比较有限,主要集中于核型相似且基因组重排较少的双翅目物种,不同昆虫科的进化模式尚不清楚。因此,Graphium属蝴蝶作为研究昆虫三维染色质结构进化模式和生物学效应的重要模型,具有深远的研究意义。
黑凤蝶(Papilio bianor) 雌虫成虫3只
美丽凤蝶(Graphium eurypylus) 雄虫成虫5只
蓝凤蝶(Graphium sarpedon) 雌虫成5只
小紫凤蝶(Graphium cloanthus) 雌虫成虫5只
研究方法:
测序方案:
二代Illumina RNA测序:5.75 ~ 7.51 Gb
二代Illumina Hi-C测序:177.29 ~415.41 Gb(293 ~ 410×)
二代Illumina ATAC测序:76 Gb(144×)
三代PacBio HIFI测序:19.76 ~ 27.18 Gb(21 ~ 38×)
分析方案:
1.基因组组装及注释
2.基因组组装质量评估:回比率、QV值、BUSCO、端粒检测
3. 比较基因组分析:进化树构建、共线性分析
4.重排位点识别
5.三维染色结果识别与比较:compartment A/B、TAD、subTAD、loop
6.ATAC-seq的peak calling与motify富集分析
7.RNA-seq的TPM定量表达分析
8. 基因编辑与qRT-PCR
对基因组进行质量评估,HiFi reads、ONT reads、NGS reads和 RNA-seq reads的整体比对率为 99.55%、99.91%、97.86%和99.1%。QV值、LAI值和BUSCO分别为56.23、15.99和99.4%(图1a)。该研究结合HiFi测序与Hi-C数据,成功生成了四种蝴蝶的染色体级别基因组组装,包括P. bianor、G. eurypylus、G. sarpedon和G. cloanthus(图1a、b)。具体基因组大小如下:P. bianor为528.52 Mb,G. eurypylus为1227.81 Mb,G. sarpedon为885.26 Mb、G. cloanthus为1107.02 Mb。最终基因组注释显示,37%至60%的序列为重复序列,36%至57%的序列为TE。利用RNA-seq注释了每个基因组约15,000个蛋白编码基因(图1a)。
在P. bianor、G. eurypylus、G. sarpedon和G. cloanthus中,约87 Mb、454 Mb、354 Mb和395 Mb的区域被注释为非功能且非重复序列,可能是由转座元件降解形成的,称为“转座元件的公墓”。这表明三种Graphium物种的基因组大小在过去1300万年中逐渐增加。TEs在基因表达和基因组大小中起重要作用,且倾向于插入在基因间区,尤其是LINEs是基因组大小扩大的主要贡献者(图1c)。
系统发育分析显示,Graphium种群在约6660万年前与P. bianor分化(图1d),这为识别基因组重排事件提供了系统发育背景。
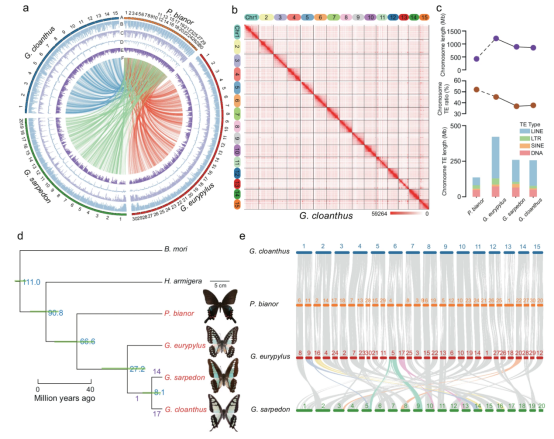
图1 四种蝴蝶的基因组进化
青凤蝶属蝴蝶基因组的剧烈重排
作者通过与P. bianor基因组的全基因组比对,分析了Graphium物种中的染色体重排事件。结果显示,G. eurypylus与P. bianor几乎没有染色体重排(图1e),而三种Graphium物种之间则存在显著的重排。以G. eurypylus为祖先核型,作者确定了G. cloanthus和G. sarpedon的重排位点(RSs),并定义了84个RSs,包括多个染色体间和染色体内融合。在G. sarpedon中,5条染色体未发生融合,其余15条由祖先染色体融合形成(图1e)。G. cloanthus则有1条未融合,其余14条均为融合结果(图1e)。尽管G. sarpedon和G. cloanthus在大约8.1百万年前分化,它们仅共享一个染色体融合位点,表明大多数融合事件是独立进化的。
蝴蝶三维染色质结构的Hi-C鉴定
作者利用Hi-C数据构建了四种蝴蝶的染色质相互作用图谱,在100 kb分辨率下,作者观察到A/B compartments,在40 kb和10 kb分辨率下分别识别了TADs和subTADs(图2a-d)。不同蝴蝶属间的A/B compartments之间的保守性(76.6%)低于三种Graphium物种之间的保守性(90%)(图3a)。
在40 kb和10 kb分辨率下,作者在P. bianor、G. eurypylus、G. sarpedon和G. cloanthus分别检测到452、1408、1031和982个TADs,以及2448、6885、4922和4577个subTADs(图2b、c和3b)。结果表明,TADs和subTADs的数量与基因组大小正相关,且P. bianor的TADs和subTADs平均长度较短(图3c)。在5 kb分辨率下,作者识别到P. bianor、G. eurypylus、G. sarpedon和G. cloanthus的loop数量分别为618、723、645和809(图2d和3d),尽管数量相似,但P. bianor的loop长度显著较短(图3d)。
最后,作者比较了三种Graphium物种之间TADs、subTADs和loops的保守性。结果显示,65–81%的lifted TADs、74–78%的lifted subTADs和52–55%的lifted loops在三种Graphium物种中保守(图3e),表明subTADs可能代表了三维染色质结构中更为保守的单元。
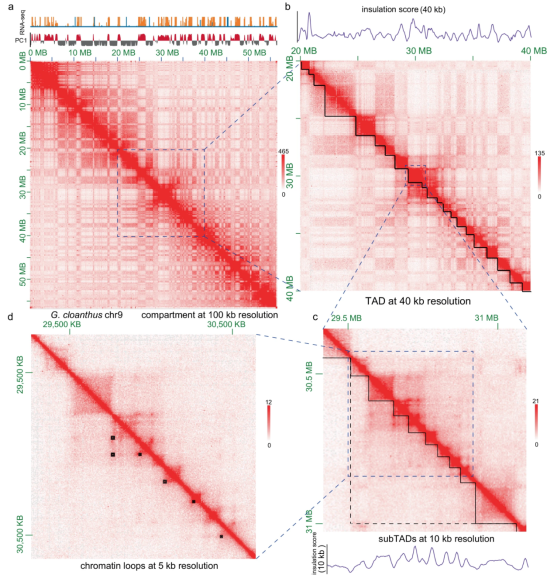
图2 Graphium cloanthus基因组的三维染色质结构
染色体间融合对三维染色质结构的影响
作者研究了G. sarpedon和G. cloanthus中染色体融合位点附近的染色质区域,发现这些区域的保守性约为90%,与整个基因组的保守性相当(图3a)。进一步分析表明,染色体重排几乎不影响染色质区域的状态,且重排位点均位于G. eurypylus的端粒或TAD边界,未观察到跨越这些区域的loop。
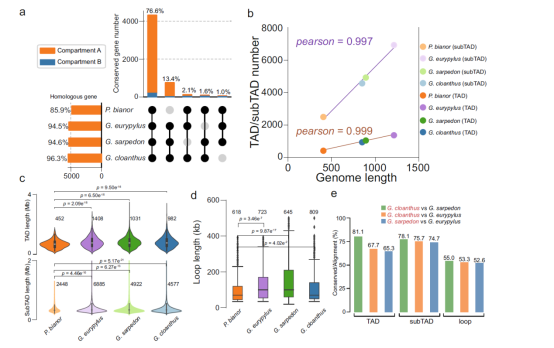
图3 三维染色质结构的保守性
在研究重排位点两侧的区域时,作者发现G. cloanthus中五个RSs的两侧区域处于不同染色质类型,未形成新TAD,而七个RSs的两侧均在compartment A ,其中两个形成了新TAD,三个形成了新subTAD(图4a)。在G. sarpedon中,八个RSs的两侧区域同样在不同染色质区域,未形成新TAD,而六个RSs的两侧区域均在compartment A ,其中五个形成新TAD,一个形成新subTAD。只有一个RS的两侧区域在compartment B,并形成了新TAD。
这些结果表明,染色体重排在compartment B之间几乎不发生,并且新TAD的形成需要相邻区域类型的一致性,尤其是在两者均为compartment A 的情况下。
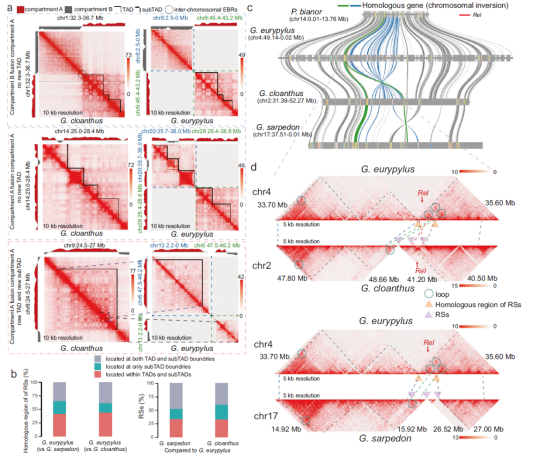
图4 重排对三维染色质结构的影响
染色体内重排对三维染色质结构的影响
作者在G. sarpedon和G. cloanthus中分别识别出66和69个100 kb以上内部染色体重排,并观察到这三种Graphium物种中TE介导的基因组扩展与TAD和subTAD的平均长度及数量显著相关(图1c,图3b,c)。比较发现,内部染色体重排对三维染色质结构的影响主要体现在大规模重排(如倒位和易位)和TE插入。研究显示,大约40.5%和43.4%的RSs分别映射到G. eurypylus的TAD和subTAD中,表明超过40%的祖先TAD和subTAD因内部重排而被打断(图4b)。
在G. sarpedon和G. cloanthus的重排基因组中,32.3%和32.1%的RSs位于TAD和subTAD内,提示这些可能是新进化的结构(图4b)。大部分大规模重排位点的侧翼区域均在compartment A ,且一些RSs在相同的染色质类型上,这表明compartment A 边界经历了更多的染色体重排事件。此外,研究发现倒位事件影响了重要基因Relish(Rel),在G. sarpedon和G. cloanthus中该基因经历了重排,导致相应的loop消失和subTAD的破坏(图4c,d)。
研究还发现,三种Graphium物种中,TE插入导致的TAD和subTAD长度变化在compartment A内较小,显示出更强的约束性,而"A到B"和"B到B"的转变则变化较大(图5b)。TE插入还形成了新的TAD,使某些compartment 从P. bianor的compartment B转变为Graphium物种的compartment A,导致levan fructotransferase基因表达上升(图5c)。这表明TE插入能够引发compartment 转换和TAD变化,从而影响基因调控。
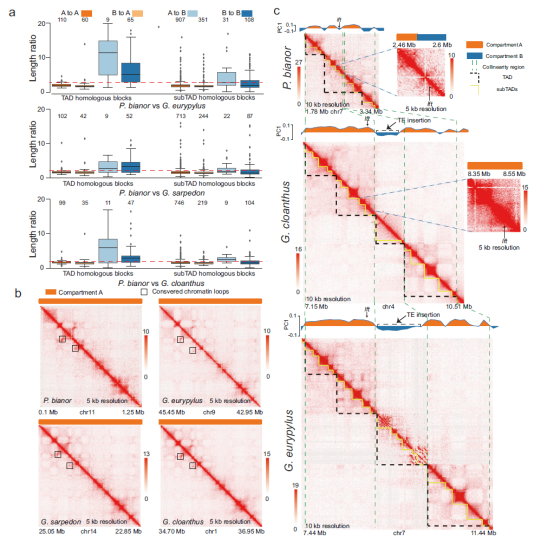
图5 TE插入改变三维染色质结构
昆虫Hox基因簇的三维染色质结构演化
作者研究了蝴蝶和果蝇中Hox基因的三维染色质结构差异。结果发现四种蝴蝶物种的Hox基因簇位于单个TAD内,而在果蝇中,由于9.6 Mb的插入,Hox基因被分为两个TAD(图6a)。蝴蝶中观察到BX-C与ANT-C位点之间有多个loops,而果蝇则没有。
通过结合Hi-C和ATAC-seq数据,作者识别了四个loops的边界绝缘体基序(图6b),并发现CTCF结合位点位于BX-C和ANT-C之间。利用CRISPR-Cas9技术生成的CTCF镶嵌型缺失突变体(mKO)表现出高死亡率和畸形,特别是在第一龄幼虫中(图6c),同时在成虫中也观察到腿部畸形和前翅缩小(图6d和6e),表明BX-C与ANT-C之间的loops对发育具有重要调控作用。
Hi-C测序结果显示,mKO突变体在缺失位点附近的交互频率显著下降,同时观察到loops的强度减弱,Antp基因表达水平下降约30%。这些结果表明Hox基因的三维染色质结构变化对蝴蝶的形态进化有重要影响。
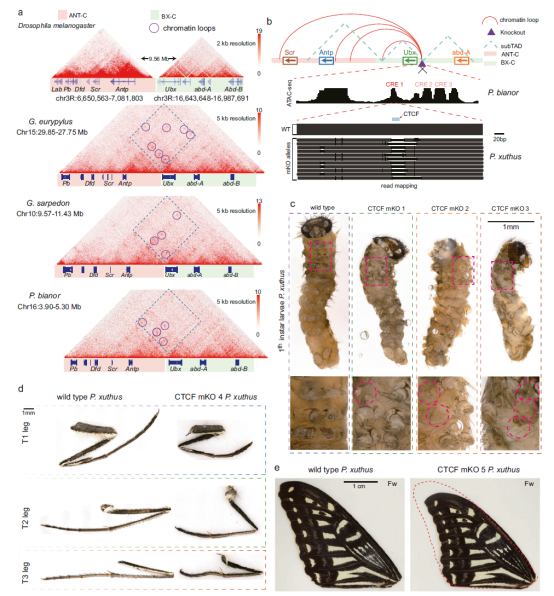
图6 CTCF中mKO的基因编辑



